-
荧光共振能量转移(fluorescence resonance energy transfer,FRET)是指供体荧光分子发射光谱与受体分子的吸收光谱有显著的重叠且分子间距小于10 nm时发生的一种非放射性的能量转移[1],导致供体荧光淬灭而受体荧光增强或不变。近些年,FRET技术以其精准高效的特点被广泛应用在分析检测领域,为检测生物分子提供了重要的分析方法。基于FRET技术,实现了活细胞内ATP分子的检测[2],金属离子如汞离子的检测[3-4],许多疾病相关基因[5-6]以及酶活性的检测等[7-8]。
银纳米簇(AgNCs),作为一种新型的低毒性“绿色”荧光标记材料,具有量子产率高、毒性低、生物相容性好等特点[9],使得其被广泛应用在多个研究领域。在银纳米簇的合成过程中,相较于其他的合成模板,DNA更具优势,如DNA具有分子识别的功能(包括对于互补链和小分子的识别),不同序列的DNA模板可调谐不同的发射波长等[10]。
研究发现,G碱基可以增强DNA/银纳米簇(DNA/AgNCs)的荧光强度[11-12],基于此,我们设计了系列非银簇模板部分的互补链,考察G碱基个数对于银簇荧光强度的影响,实验结果显示,暴露的G碱基个数与银簇的荧光强度呈正相关关系。该实验不仅验证了G碱基对于银簇荧光的增强作用,还提示我们在设计含有G-四联体适配体的荧光探针时,可通过改变非银簇部分的互补链长短来调控荧光的淬灭及恢复程度,以获得最佳的检测效果。
-
VS-100C恒温混匀仪(无锡沃信仪器制造有限公司);FL-6500荧光分光光度计(PerkinElmer);ZEN3600粒径电位测定仪(英国马尔文公司);精密电子天平(北京赛多利斯仪器系统有限公司);Vortex-Genie2多功能旋涡混合器(美国Scientific Industries公司);TGL-16C离心机(上海安亭科学仪器厂);实验室pH计 FE20[梅特勒-托利多仪器(上海)有限公司];Tecnai G2 F30高分辨率电子显微镜(荷兰FEI公司)
-
硝酸银、硼氢化钠、盐酸(国药集团化学试剂有限公司);三羟甲基氨基甲烷(Tris,大连美仑生物技术有限公司);氯化镁、氯化钠、氯化钾(上海泰坦科技股份有限公司);试剂均为分析纯。实验用水为屈臣氏蒸馏水。
相关DNA序列由生工生物工程(上海)股份有限公司合成。合成DNA-AgNCs的模板序列及P1A5C5的互补序列如表1、表2所示。
表 1 DNA/AgNCs的模板序列
名称 序列(5′—3′) P1C5 GGAGGTGGTGGGGCCCCCTAATTCCCCC P1AC5 GGAGGTGGTGGGGACCCCCTAATTCCCCC P1A5C5 GGAGGTGGTGGGGAAAAACCCCCTAATTCCCCC P1N GGAGGTGGTGGGGCCCTAACTCCCC P1Y GGAGGTGGTGGGGCCCTTAATCCCC 表 2 P1A5C5的互补序列
名称 序列(5′—3′) 0G CCTCCACCACCCCTTTTT 1G CTCCACCACCCCTTTTT 2G TCCACCACCCCTTTTT 4G TCCACCCCTTTTT 5G CACCCCTTTTT 6G ACCCCTTTTT 7G CCCTTTTT -
加入相应体积的20 mmol/L Tris-HCl(pH=7.4)缓冲溶液将DNA溶解,即制得100μmol/L DNA溶液,将制得的DNA溶液95 ℃加热5 min后,冰水浴冷却10 min。
-
参考文献中的合成方法[13],将一定体积的硝酸银溶液加入到上述DNA溶液中(20 mmol/L Tris-HCl,pH=7.4),充分震荡混匀,25 ℃孵育20 min,静置,将一定体积新配制的硼氢化钠引入到上述反应混合物中,最终使得体系中DNA、硝酸银、硼氢化钠的浓度分别为5、30、30μmol/L(即DNA:Ag+∶NaBH4的摩尔比为1∶6∶6),剧烈震荡混匀,室温下避光反应3 h后,4 ℃避光反应过夜。得到的银纳米簇溶液在4 ℃保存以备用。
-
荧光图谱表征:将2μmol/L的银纳米簇溶液在200~800 nm波长范围内进行荧光光谱预扫描,设置激发和发射狭缝宽度为10 nm,扫描速度为1200 nm/min,电压值为400 V。
高分辨率透射电子显微镜表征:将银纳米簇溶液滴加铜网后观察。
-
在含有100 mmol/L NaCl,2 mmol/L MgCl2,5 mmol/L KCl的20 mmol/L Tris-HCl缓冲溶液中(pH=7.4),加入银簇溶液(2μmol/L),将互补的DNA溶液(表2)以1∶1的摩尔比分别加入到上述体系溶液中,充分震荡混匀,37 ℃孵育20 min。在室温条件下进行荧光光谱测量。激发波长为486 nm,激发和发射狭缝宽度为10 nm,扫描速度为1200 nm/min,电压值为400 V。
-
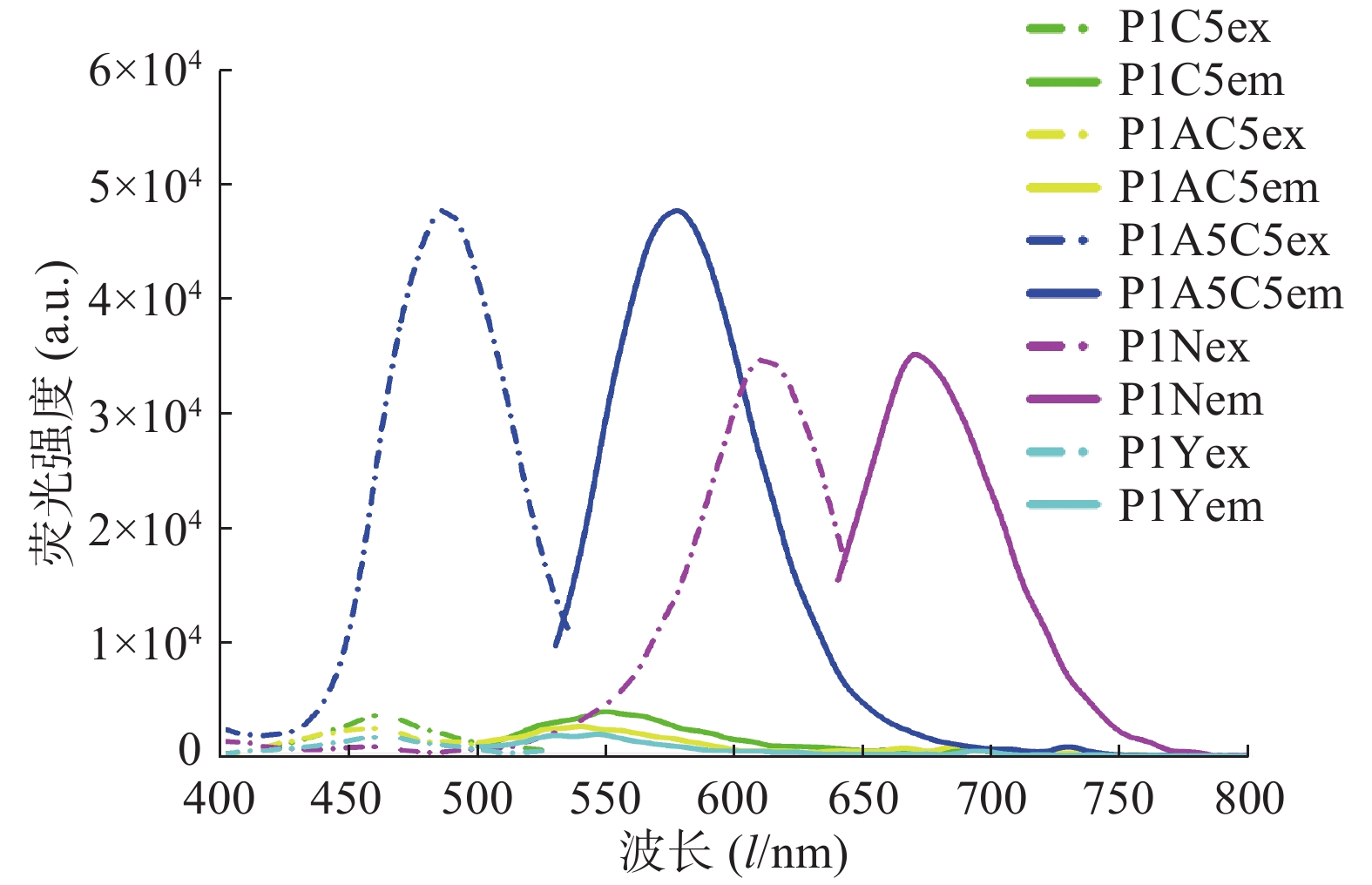
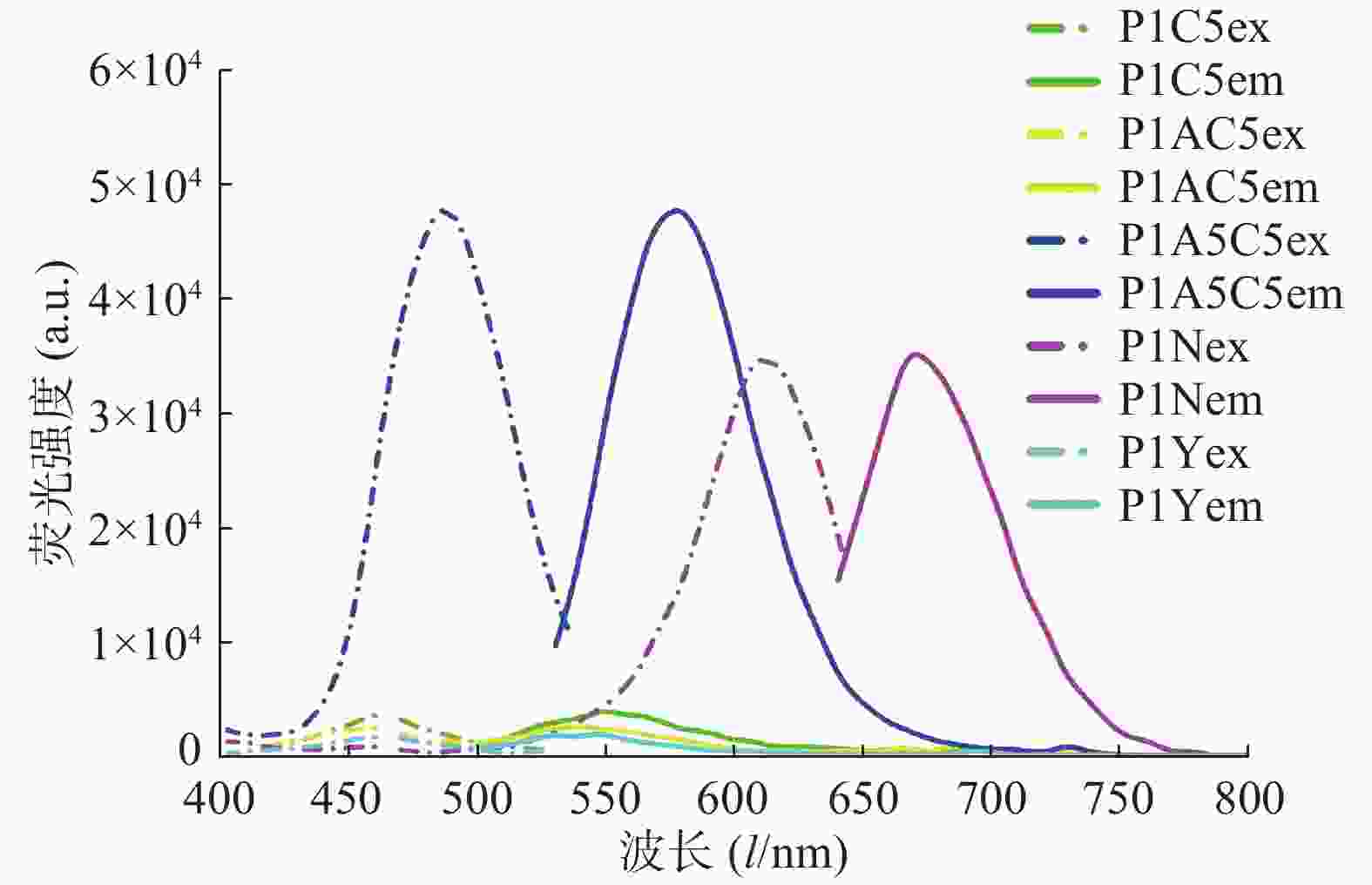
在进行DNA/AgNCs的合成时,基于胞嘧啶碱基与银离子的作用,一般选择富含胞嘧啶碱基的序列作为合成模板,笔者根据文献报道的模板序列结合模板优化设计[13-16],选择了5种银簇模板并进行了相应的碱基优化设计,如表1所示,结果显示P1A5C5的荧光信号强度较高(如图1),且4 ℃避光保存45 d后,荧光信号强度基本不变,稳定性良好。因此,我们使用该序列合成的DNA/AgNCs进行后续的实验研究。
对生成的P1A5C5银纳米簇体系进行荧光激发光谱和发射光谱的表征,结果如图2所示,在487 nm激发条件下,发射波长为577 nm。
DNA/AgNCs的高分辨透射电子显微镜(HRTEM)图片如图3所示,从图中可以看出,DNA/AgNCs的直径约为2~3 nm,分散性良好。
-
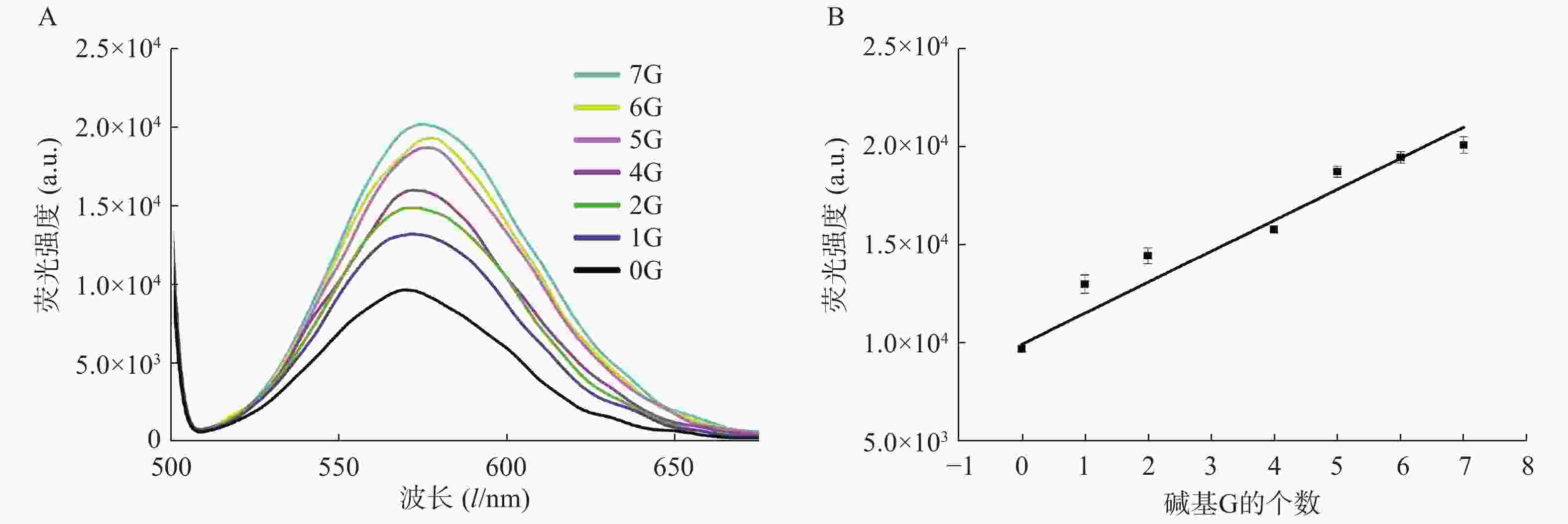
在银簇P1A5C5序列5′-GGAGGTGGTGGGGAAAAACCCCCTAATTCCCCC-3′中,CCCCCTAATTCCCCC为成簇模板序列,下划线部分为岩沙海葵毒素的G-四联体适配体[17]。基于G碱基对银簇的荧光信号强度有增强作用这一现象,我们设计了一系列适配体部分的互补序列(表2),通过C-G碱基互补,适配体部分暴露的G碱基个数不同,导致与银纳米簇作用的碱基G数目不同,进而对银纳米簇的荧光信号强度产生不同的影响。结果显示,随着暴露的G碱基个数的增加,荧光信号强度增加(图4A),对暴露的G碱基个数与荧光信号强度关系进行拟合,得到的线性方程为:Y=1726.1X+8972.5,r=0.9789(图4 B)。该研究证明了G碱基对银簇的荧光具有增强效果,反之,通过C-G碱基互补配对,G碱基与银簇的作用位点被占据,无法与银簇作用,难以达到增强荧光信号强度的作用。因此,随着暴露碱基个数的减少,荧光强度减弱。基于此,可实现与G-四联体适配体有关的荧光开关的设计,进而实现对目标物的检测。
在DNA序列和银纳米簇进行碱基互补配对时,结合互补序列的Tm值,我们研究了互补链间实现碱基互补配对所需的温度和时间。实验结果显示,相较于4 ℃,在37 ℃条件下孵育,互补链间相互作用较强,容易实现碱基互补配对,荧光信号强度变化较为明显;同时,考察了在37 ℃下作用1 h内的荧光变化情况,结果显示,荧光变化强度随时间未发生明显变化,最终选择20 min作为孵育时间。
-
该实验利用含有G四联体适配体的DNA序列合成了荧光银纳米簇,基于G碱基可以增强银纳米簇的荧光这一现象,并结合碱基互补配对的原则,设计了8种G四联体适配体的互补链,在互补链和适配体部分碱基互补配对后,荧光信号强度也产生相应的变化。随着互补链长度的缩短,暴露的G碱基个数增加,荧光信号增强,且暴露的G碱基个数与荧光信号强度拟合得到的线性方程为:Y=1726.1X+8972.5,r=0.9789。该实验不仅证实了G碱基对荧光银纳米簇有荧光增强作用,还提示我们在设计含有G四联体的银纳米簇荧光探针时,可以通过互补链对荧光信号的干扰程度来调控荧光的开闭,这对进一步扩展银纳米簇在荧光分析方法中的应用具有指导意义。
The effect of different guanine base number on fluorescence intensity of DNA/ silver nanoclusters
-
摘要:
目的 通过C-G碱基互补配对的方式,考察不同鸟嘌呤碱基(G)数目对DNA/银纳米簇荧光信号强度的影响,以此来探究新的荧光探针开关构建的方法。 方法 利用核酸碱基互补配对原则,设计了一系列银纳米簇的适配体部分的互补序列,考察了银纳米簇的适配体序列中暴露的G碱基个数对荧光信号的影响。 结果 碱基G可增强银纳米簇的荧光信号强度,且荧光信号强度与G碱基个数呈现正相关关系,拟合线性方程为Y=1726.1X+8972.5,r=0.9789。 结论 该实验研究对于调节银纳米簇的荧光强度以及设计适配体为G四联体的荧光探针开关具有借鉴意义。 Abstract:Objective To investigate the effect of different guanine base numbers on the fluorescence intensity of DNA/ silver nanoclusters through C-G base complementary pairing, in order to explore a new method for the construction of fluorescent probe switches. Methods Designed complementary sequences of aptamer parts of a series of silver nanoclusters by using the nucleic acid base complementary pairing principle, and investigated the effect of the number of G bases exposed in the aptamer sequence on the fluorescence signal. Results Base G could enhance the fluorescence signal intensity of silver nanoclusters, and the fluorescence signal strength was positively correlated with the number of G bases. The fitting linear equation was Y=1726.1X+8972.5, r=0.9789. Conclusion This study is a great reference for the regulation of fluorescence intensity of silver nanoclusters and the design of G quadruplet aptamer fluorescent probe switch. -
Key words:
- DNA/AgNCs /
- complementary pairing /
- fluorescence probe /
- aptamer
-
表 1 DNA/AgNCs的模板序列
名称 序列(5′—3′) P1C5 GGAGGTGGTGGGGCCCCCTAATTCCCCC P1AC5 GGAGGTGGTGGGGACCCCCTAATTCCCCC P1A5C5 GGAGGTGGTGGGGAAAAACCCCCTAATTCCCCC P1N GGAGGTGGTGGGGCCCTAACTCCCC P1Y GGAGGTGGTGGGGCCCTTAATCCCC 表 2 P1A5C5的互补序列
名称 序列(5′—3′) 0G CCTCCACCACCCCTTTTT 1G CTCCACCACCCCTTTTT 2G TCCACCACCCCTTTTT 4G TCCACCCCTTTTT 5G CACCCCTTTTT 6G ACCCCTTTTT 7G CCCTTTTT -
[1] CHEN X, KO S K, KIM M J, et al. A thiol-specific fluorescent probe and its application for bioimaging[J]. Chem Commun (Camb),2010,46(16):2751-2753. doi: 10.1039/b925453f [2] ZHAO J, GAO J, XUE W, et al. Upconversion luminescence-activated DNA nanodevice for ATP sensing in living cells[J]. J Am Chem Soc,2018,140(2):578-581. doi: 10.1021/jacs.7b11161 [3] AMIRI S, AHMADI R, SALIMI A, et al. Ultrasensitive and highly selective FRET aptasensor for Hg2+ measurement in fish samples using carbon dots/AuNPs as donor/acceptor platform[J]. New J Chem,2018,42(19):16027-16035. doi: 10.1039/C8NJ02781A [4] GUO H, LI J S, LI Y W, et al. A turn-on fluorescent sensor for Hg2+ detection based on graphene oxide and DNA aptamers[J]. New J Chem,2018,42(13):11147-11152. doi: 10.1039/C8NJ01709C [5] FANG B Y, LI C, AN J, et al. HIV-related DNA detection through switching on hybridized quenched fluorescent DNA-Ag nanoclusters[J]. Nanoscale,2018,10(12):5532-5538. doi: 10.1039/C7NR09647J [6] GUO W, YUAN J, DONG Q, et al. Highly sequence-dependent formation of fluorescent silver nanoclusters in hybridized DNA duplexes for single nucleotide mutation identification[J]. J Am Chem Soc,2010,132(3):932-934. doi: 10.1021/ja907075s [7] WANG L J, REN M, ZHANG Q Y, et al. Excision repair-initiated enzyme-assisted bicyclic cascade signal amplification for ultrasensitive detection of uracil-DNA glycosylase[J]. Anal Chem,2017,89(8):4488-4494. doi: 10.1021/acs.analchem.6b04673 [8] XU N, WANG Q, LEI J, et al. Label-free triple-helix aptamer as sensing platform for “signal-on” fluorescent detection of thrombin[J]. Talanta,2015,132:387-391. doi: 10.1016/j.talanta.2014.09.031 [9] LATORRE A, SOMOZA Á. DNA-mediated silver nanoclusters: synthesis, properties and applications[J]. Chembiochem,2012,13(7):951-958. doi: 10.1002/cbic.201200053 [10] LIU J W. DNA-stabilized, fluorescent, metal nanoclusters for biosensor development[J]. Trac Trends Anal Chem,2014,58:99-111. doi: 10.1016/j.trac.2013.12.014 [11] YEH H C, SHARMA J, HAN J J, et al. A DNA-silver nanocluster probe that fluoresces upon hybridization[J]. Nano Lett,2010,10(8):3106-3110. doi: 10.1021/nl101773c [12] WALCZAK S, MORISHITA K, AHMED M, et al. Towards understanding of poly-guanine activated fluorescent silver nanoclusters[J]. Nanotechnology,2014,25(15):155501. doi: 10.1088/0957-4484/25/15/155501 [13] RICHARDS C I, CHOI S, HSIANG J C, et al. Oligonucleotide-stabilized Ag nanocluster fluorophores[J]. J Am Chem Soc,2008,130(15):5038-5039. doi: 10.1021/ja8005644 [14] MA J L, YIN B C, YE B C. DNA template-regulated intergrowth of a fluorescent silver nanocluster emitter pair[J]. RSC Adv,2015,5(119):98467-98471. doi: 10.1039/C5RA21159J [15] LIN R, TAO G, CHEN Y, et al. Constructing a robust fluorescent DNA-stabilized silver nanocluster probe module by attaching a duplex moiety[J]. Chemistry,2017,23(45):10893-10900. doi: 10.1002/chem.201701879 [16] JIANG Y T, TANG Y G, MIAO P. Polydopamine nanosphere@silver nanoclusters for fluorescence detection of multiplex tumor markers[J]. Nanoscale,2019,11(17):8119-8123. doi: 10.1039/C9NR01307E [17] GAO S, ZHENG X, HU B, et al. Enzyme-linked, aptamer-based, competitive biolayer interferometry biosensor for palytoxin[J]. Biosens Bioelectron,2017,89(pt 2):952-958. -



 下载:
下载: